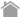miRNA-mRNAペアリング解析
(RIP-Assay Kit for microRNA)
近年、miRNAは発生・分化・細胞増殖・アポトーシスなどの様々なプロセスに関与することが報告されてきました。さらに、組織や疾患特異的な発現および発現量の増減が報告されたことから、miRNAのターゲットmRNAの同定は、疾患メカニズムの解明だけでなく、診断や創薬への応用にも重要だと考えられています。しかしながら、miRNAのmRNAへの結合にはミスマッチが含まれることや、標的mRNAの認識に最も重要とされる5'末端から2-8番目の塩基配列(seed sequence)が7塩基と非常に短いことから、1つのmiRNAは複数のmRNAの翻訳制御に関与していると考えられており、miRNAの標的mRNAの同定は困難とされてきました。これまでに、コンピューター(in silico)でのターゲットmRNA予測方法が開発されてきましたが、精度の問題上、完全な方法とは言えず、生化学的手法によるターゲット探索方法の開発が求められてきました。
RIP-Assay Kit for microRNA(RN1005)は、同一のRNP複合体中のlarge RNAとsmall RNAの両方を回収できるキットです。目的のmiRNAを遺伝子導入した後、抗AGO2抗体を用いてRIP-Assayを行い、続いて得られたsmall/large RNAについてマイクロアレイ解析を行うことで、生化学的手法によりmiRNAの標的mRNAを効率よく同定することが可能になります。
材料・方法
miRNA: let-7a (2本鎖miRNA、3' 突出末端)
細胞: HEK293T
抗体: Anti-EIF2C2 (AGO2) (Human) mAb
(RN003M)、Mouse IgG2a (M076-3)
キット:RIP-Assay Kit for microRNA (RN1005)
- 1. let-7a-1配列をもとにlet-7a duplexを合成
(IDT社) - 2. HEK293T細胞へlet-7aを導入して、24時間後に
RIP-Assay実施 - 3. Large RNAおよびsmall RNAのマイクロアレイ解析
(アジレント社チップ)
mRNA: Whole Human Genome 4x44k
miRNA: Human miRNAマイクロアレイキット8x15k - 4. ルシフェラーゼアッセイでの翻訳抑制確認
実験の目的
RIP-Assay Kit for microRNAを用いたmiRNA-mRNAペアリング解析の実証例を得るため、let-7aの標的mRNAの探索を行いました。
let-7は、線虫において最初に同定されたmiRNAです。let-7は種を超えてよく保存されており、ヒトではlet-7ファミリーとして10種類のmiRNAの存在が知られています。これまでに、癌におけるlet-7ファミリーの発現の増減が報告されており、そのうちの一つのlet-7aは、肺癌、肝細胞癌、腎臓癌、膵臓癌など多くの癌で発現低下が認められています。let-7aの異常な発現低下により癌遺伝子のRASやHMGA2が過剰発現することも明らかになっていることから、let-7aをモデル分子として選択しました。
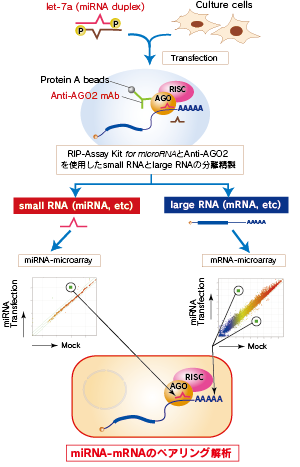
miRNAのマイクロアレイ解析では、let-7a導入後24時間で、total RNA中のFold Change (FC)が98倍以上、AGO2-RIP RNAでは6倍以上のmiRNAから、let-7aが検出されました。アレイデータでは、let-7a導入により、他のlet-7ファミリー分子も検出されていますが、これらはlet-7 ファミリー間の配列相同性の高さに起因するcross-hybridizationによるものと考えられます。Total RNAだけでなく、AGO2-RIPでもlet-7aが検出されたことから、導入したlet-7aが細胞内のRISCに効率よく取り込まれていることを確認できました(図1上段)。
mRNAのマイクロアレイ解析では、興味深いデータが示されています。Total RNAの解析では、let-7aを過剰導入しているにも拘らず、ほとんどのmRNAの発現量に変化が認められませんでした。つまり、let-7aが過剰発現したとしても、mRNAの発現プロファイルにはほとんど影響しないことが示されています。一方、AGO2-RIP RNA中のmRNA発現プロファイルは大きく変化しており、Gene-X、Y、ZなどはFCが6倍以上の値を示しました(図1下段)。let-7aの増加に伴い、トランスクリプトームレベルでの変化は生じないが、AGO2との結合状態が大きく変動するmRNAが多く認められたということは、miRNAが転写レベルではなく翻訳レベルでmRNAの発現を制御しているというモデルを支持する結果となりました。
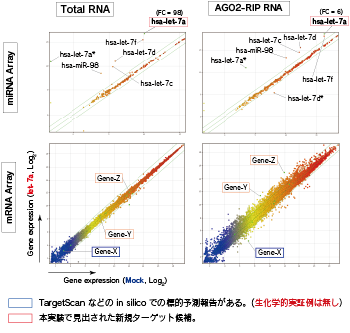
 図1. Let-7a導入によるmRNA結合状態変化
図1. Let-7a導入によるmRNA結合状態変化
(アレイデータ)
上の2つは、total RNAから得られたmiRNA(左)およびAGO2-RIPにより得られたmiRNA(右)のスキャッタープロットを示します。同様に、下の2つは、total RNAから得られたmRNA(左)およびAGO2-RIPにより得られたmRNA(右)のスキャッタープロットです。縦軸にlet-7aを導入したサンプル、横軸はmockをとり、let-7a導入による発現変動および結合量の変化を表しています。miRNAアレイのスキャッタープロット中に示したmiRNAは、total RNAではFCが98倍以上、AGO2-RIPではFCが6倍以上変動したmiRNAです。mRNAアレイデータで示したGene-X、Y、Zは、AGO2-RIPにおいて6倍以上の結合量の変化が認められました。
Gene-Xは、TargetScanなどのin silicoでの標的予測報告はされていましたが、生化学的実証例はありませんでした。また、Gene-Y、Zは標的予測報告のない遺伝子でした。let-7aのseed sequenceとの相補性を指標としてこれら3つの遺伝子の標的配列部位を調べると、それぞれ、3'-UTRやCDRに含まれることから、この部分をクローニングしてレポーターベクターに挿入し、実際にlet-7a導入による翻訳抑制が認められるのか検証しました(図2)。その結果、Gene-Xでは、let-7a導入により有意にルシフェラーゼ活性が抑制されました。Gene-Xは、let-7a導入後のAGO2-RIPではAGO2との結合量が低下していますがルシフェラーゼアッセイでの抑制効果が認められたことから、Gene-Xは分解速度が早いためAGO2との結合量が低下したことが考えられました。以上の結果から、AGO2-RIP-Chipにより、let-7の新規のターゲットmRNAとしてGene-Xを見出すことができました。
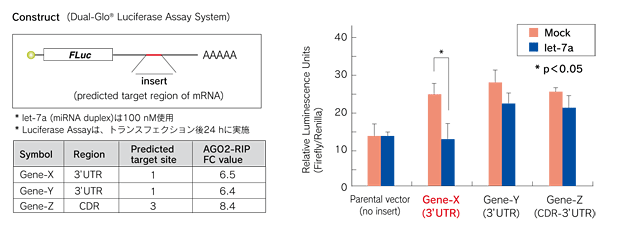
図2. HEK293T細胞でのLucレポーターアッセイ(n=3)
表はGene-X、Y、Zのlet-7a相補配列の位置および数を示します。Lucレポーターアッセイに使用するベクターは、ホタルルシフェラーゼのコード領域直下に表で示したGene-X、Y、Zのlet-7a相補配列を含む領域を挿入しています。ベクターおよびlet-7aを導入して24時間後に細胞を回収してルシフェラーゼ活性を測定しました。グラフの縦軸は、ルシフェラーゼの測定値をRenillaのルシフェラーゼ活性測定値で割って標準化した値です。Gene-Xについて、let-7a導入によるルシフェラーゼ活性の低下が認められました。