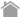蛍光タンパク質を用いたタンパク質間相互作用検出技術 Fluoppi の開発
株式会社医学生物学研究所
研究開発本部 LSTR 開発ユニット
創薬技術開発プロジェクト
グループリーダー 渡部 拓
1.蛍光タンパク質の特徴
1962年、下村脩らは「日の光の下では僅かに緑色であり、タングステン光では黄色、そして、紫外線の下では非常に明るい緑色蛍光を発するタンパク質が単離された。」と述べ、オワンクラゲ(Aquorea victoria )から緑色蛍光タンパク質(GFP)を発見したことを報告した[1]。その後タンパク質レベルでの解析を経て、1992年にGFP遺伝子がクローニングされ、1994年には、線虫および大腸菌でのGFPタンパク質発現が報告された[2-4]。これにより、GFP遺伝子は発色団形成に必要な全ての情報をアミノ酸配列上に保有しており、オワンクラゲ特有の酵素を必要とすることなく、単独で蛍光性を獲得可能であることが証明された[5]。すなわち、GFPにより、様々な生物を生きたままの状態で光らせることが可能であることが示された。その後、GFPの変異体が次々と報告され、Cyan fluorescent protein (CFP)やYelow fluorescent protein (YFP)など蛍光特性の多様性が拡張された[5-7]。
1999年、Matzらは花虫類(Anthozoa species)から6つの蛍光タンパク質をクローニングしたことを報告した[8]。その内の一つが、蛍光波長の極大が583 nmを示すDsRedである。赤色蛍光タンパク質の登場は、GFPと組み合わせることで複数の細胞内イベントを同時に解析することが容易となるため、その応用に期待が高まった。しかしながら、DsRedはそれ自身で恒常的(obligate)に四量体を形成するため、観察対象のタンパク質に直接融合して細胞内の局在を解析する用途には不向きであることが予想された[9]。それに対し、CampbellらはDsRedに33か所のアミノ酸変異を導入した単量体のmRFP1を報告し、以後、タンパク質の局在解析には単量体蛍光タンパク質を使用することが主流となった[10, 11]。mRFP1は後に更に進化を遂げ、蛍光波長の多様性が拡大した[12]。
蛍光タンパク質の応用は、解析対象に直接融合し、生きた細胞でその局在を観察する方法だけに留まらない。例えば、GFPの立体構造形成が蛍光の有無を指標に測定できる点を利用し、アルツハイマー病治療薬を狙ったAmyloyd β (Aβ)ペプチド凝集阻害薬を探索する方法や、蛍光タンパク質が細胞内で迅速に分解される点を利用し、細胞周期特異的に蛍光タンパク質を分解させることで細胞周期を可視化する方法(Fluorescent Ubiqutination-based Cell Cycle Indicator: Fucci)、mRFPとEGFPのpH耐性の差もしくは、Keima-Redの励起波長がpH依存的に変化する点を利用し、オートファジーを可視化する方法などが挙げられる[13-16]。また、蛍光の不可逆的褪色を利用した、Fluorescence recovery after photobleaching (FRAP)やFluorescence loss in photobleaching (FLIP)、不可逆的光活性化が可能なPhotoactivatable GFP (PA-GFP)、不可逆的光変換が可能なKaede、可逆的光変換型のDronpaを用いて、細胞内タンパク質の拡散速度の測定や、特定の細胞のみをハイライトする実験にも利用されている[17-20]。この様に蛍光タンパク質の特徴を活用した技術は多数報告されている。
2.蛍光タンパク質の特徴を活用したタンパク質間相互作用検出法
タンパク質間相互作用(Potein protein interaction: PPI)はシグナル伝達の根幹であり、細胞の構造維持や機能発揮に必要不可欠である。また、がんをはじめとした幅広い疾患領域において重要な役割を果たしているため、現在、創薬標的として活発に阻害薬や誘導剤、安定化剤の開発が行われている[21-23]。細胞内PPIを標的としたPPI調整剤は、試験管内での活性に加え細胞内での活性評価が重要と考えられる。というのも、①in vitro の生化学アッセイでは、物理的・非特異的にPPIを解離させるような化合物が存在するため、また、②PPI調整剤は分子量が大きい傾向にあり、細胞膜透過性が低いことが予想されるため、更に、③細胞内は多様な物質が高密度に集積した場であり、その様な複雑な環境下で活性を有する必要があるため、である。近年、Voglerらは、文献報告のあった6種のBcl2阻害剤を比較し、細胞内で実際にBcl2に作用する化合物は1種のみであり、他5種は意図した作用機序とは異なるメカニズムで細胞死を引き起こすことを示した[24]。候補となるPPI調整剤を早い段階で評価するためには、同化合物が細胞内で目的の活性を有するか否か、すなわちPPIを調整する効果を有するか否かを評価する必要があると考えられる。
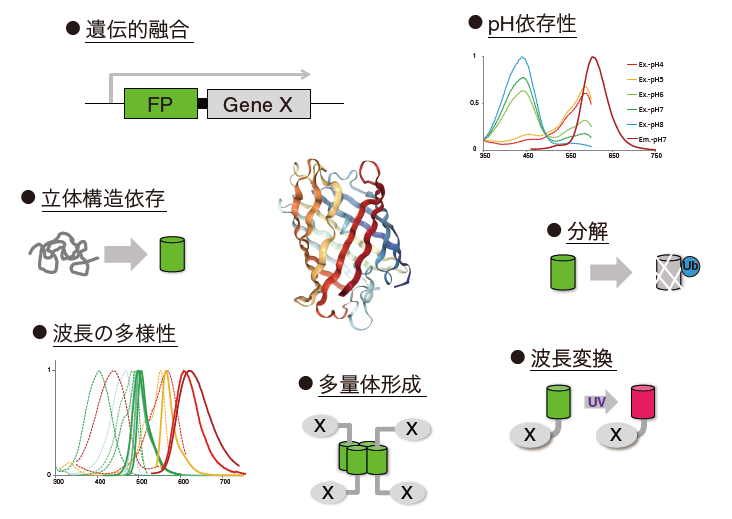
それでは、蛍光タンパク質の特徴を活用したPPI検出方法にはどの様なものがあるのだろうか。私たちは、同手法は大きく2群に分類することができると考える。「第一群」は蛍光タンパク質などのタグの近接を利用した手法であり、解析対象のタンパク質XとYに遺伝的に融合したタグ同士が近接することで、蛍光強度が変化するタイプである。具体的には、FRET (Fluorescence resonance energy transfer) [25 -29]、PCA (Protein complementation assay)を蛍光タンパク質で実施したBiFC (Bimolecular fluorescence complementation) [30, 31]、ddFP (Dimerization-dependent fluorescent protein)を用いたFPX (Fluorescent protein exchange) [32, 33]が挙げられる。これらの手法ではタグ同士が物理的に近接する必要があるため一般にXとYの末端間の距離が短いことが必要とされ、更に、解析対象とタグを接続するペプチドリンカーの最適化が求められる。「第二群」は蛍光タンパク質の再分布(redistribution)を利用したタイプである。蛍光タンパク質を融合した解析対象の細胞内局在(分布)がPPIの有無で変化することを検出する。Xには局在化させるためのタグ(局在化タグ)を、Yには蛍光タンパク質をそれぞれ遺伝的に融合し、Yが局在化タグと同じ場所に集積するか否かによりPPIを判定する手法[34-38]や、多価の粒子を利用してXとYの相互作用および特定の化合物の添加依存的にクラスターを形成させる手法[39]が挙げられる。前者は局在化させる場所によってはバックグラウンドが高いという課題があり、後者は不可逆的であると言われている。しかし、これらの手法はハイコンテント解析(High content analysis: HCA)機器や解析プログラムの発展により、近年創薬研究分野にて注目を浴びている[34]。
3.Phase separationを利用したタンパク質間相互作用検出法 (Fluoppi)
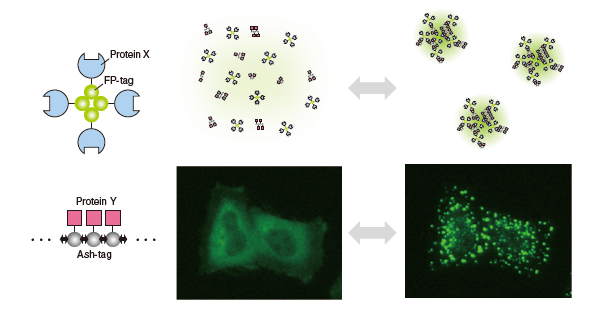
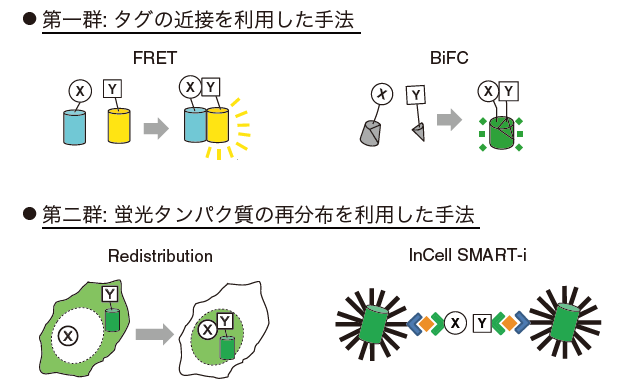
当研究グループでは、細胞内でPPIを検出する新たな手法の開発を目指し、「第二群」に属するFluoppiを開発した。開発時に着目したのは、細胞内タンパク質が相転移により液状を維持しつつ分離され、区画化される現象(Liquid phase separation)である。Phase separationによるPhase separated dropletは、細胞内に普遍的に存在する膜に包まれないオルガネラ様構造体として近年研究が進んでいる[40, 41]。同構造体の多くはProcessing body (P-body)や核小体などのRNA-タンパク質の複合体(RNP body)であるが、近年、Liらはシグナル伝達因子の多価結合がPhase separated dropletを形成することを実証した[42]。この研究にヒントを得て、当研究グループではホモ多量化能を有する2つのタンパク質タグを採用した。1つ目のタンパク質タグは、p62/SQSTM1のPB1ドメインである(製品名はAssembly Helper Tag (Ash-Tag)としている)。 p62のPB1ドメインは、一方の側に酸性/疎水性残基を含み、他方の側にリジンおよびアルギニン残基を含有する。これにより、前-背面のトポロジーで高分子量のホモオリゴマーを形成する[43, 44]。第2のタグは、obligate四量体を形成するサンゴ由来の緑色蛍光タンパク質、Azami-Green (AG)である[45]。これらのタグを利用し、生細胞内における蛍光輝点として、PPI依存的にPhase separated dropletを形成させることができた。これまで、蛍光タンパク質はどれだけ単量体であるか(どれだけ多量体形成能が低いか)、という点に着目されてきたが、私たちは視点を変え、蛍光タンパク質の四量体をむしろ活用することで、細胞内Liquid phase separationの原理を利用したPPI検出方法へ応用することに成功した。Fluoppiと命名したこの方法は、HCAおよびHigh Throughput Screening (HTS)に適しており、多くの異なるPPIを生きた細胞内でモニタリングすることができる。詳細な技術内容については参考文献46(PMID: 28406179)を参照頂けると幸いである。
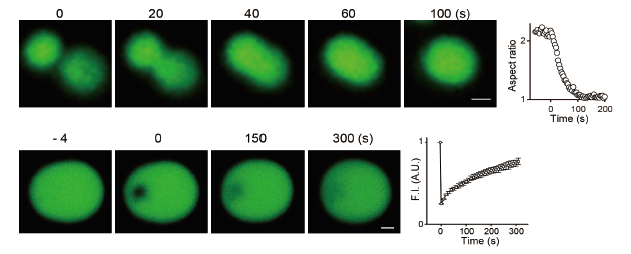
Fluoppi で形成されたLiquid phase droplet
(上) 二つのdropletが融合する様子。グラフのAspect ratioはdropletの長径と短径の比を示す。時間経過とともに1に近づき、融合後、球形となることがわかる。
(下) Dropletの一部を褪色させ、その領域の蛍光回復を測定した結果を示す。Droplet内部が流動的であることがわかる。
参考文献
- Shimomura O, et al. Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. Journal of cellular and comparative physiology, 59: 223-239, 1962
- Prasher DC, et al. Primary structure of the Aequorea victoria green-fluorescent protein. Gene 111: 229-233, 1992
- Chalfie M, et al. Green fluorescent protein as a marker for gene expression. Science (New York, N.Y.), 263: 802-805, 1994
- Inouye S and Tsuji FI, Aequorea green fluorescent protein. Expression of the gene and fluorescence characteristics of the recombinant protein. FEBS letters, 341: 277-280, 1994
- Tsien RY, The green fluorescent protein. Annual review of biochemistry, 67: 509-544, 1998
- Heim R, et al. Wavelength mutations and posttranslational autoxidation of green fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America, 91: 12501-12504, 1994
- Cubitt AB, et al. Understanding, improving and using green fluorescent proteins. Trends in biochemical sciences, 20: 448-455, 1995
- Matz MV, et al. Fluorescent proteins from nonbioluminescent Anthozoa species. Nature biotechnology, 17: 969-973, 1999
- Baird GS, et al. Biochemistry, mutagenesis, and oligomerization of DsRed, a red fluorescent protein from coral. Proceedings of the National Academy of Sciences of the United States of America, 97: 11984-11989, 2000
- Campbell RE, et al. A monomeric red fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America, 99: 7877-7882, 2002
- Zacharias DA, et al. Partitioning of lipid-modified monomeric GFPs into membrane microdomains of live cells. Science (New York, N.Y.), 296: 913-916, 2002
- Shaner NC, et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nature biotechnology, 22: 1567-1572, 2004
- Kim W, et al. A high-throughput screen for compounds that inhibit aggregation of the Alzheimer's peptide. ACS chemical biology, 1: 461-469, 2006
- Sakaue-Sawano A, et al. Visualizing spatiotemporal dynamics of multicellular cell-cycle progression. Cell, 132: 487-498, 2008
- Kimura S, et al. Dissection of the autophagosome maturation process by a novel reporter protein, tandem fluorescent-tagged LC3. Autophagy, 3: 452-460, 2007
- Katayama H, et al. A sensitive and quantitative technique for detecting autophagic events based on lysosomal delivery. Chemistry & biology, 18: 1042-1052, 2011
- Swaminathan R, et al. Photobleaching recovery and anisotropy decay of green fluorescent protein GFP-S65T in solution and cells: cytoplasmic viscosity probed by green fluorescent protein translational and rotational diffusion. Biophysical journal, 72: 1900-1907, 1997
- White J and Stelzer E, Photobleaching GFP reveals protein dynamics inside live cells. Trends Cell Biol, 9: 61-65, 1999
- Ando R, et al. An optical marker based on the UV-induced green-to-red photoconversion of a fluorescent protein. Proceedings of the National Academy of Sciences of the United States of America, 99: 12651-12656, 2002
- Ando R, et al. Regulated fast nucleocytoplasmic shuttling observed by reversible protein highlighting. Science, 306: 1370-1373, 2004
- Fry DC, Protein-protein interactions as targets for small molecule drug discovery. Biopolymers, 84: 535-552, 2006
- Arkin M. R, et al. Small-molecule inhibitors of protein-protein interactions: progressing toward the reality. Chemistry & biology, 21: 1102-1114, 2014
- Fischer ES, et al. SPLINTS: small-molecule protein ligand interface stabilizers. Current opinion in structural biology, 37: 115-122, 2016
- Vogler M, et al. Different forms of cell death induced by putative BCL2 inhibitors. Cell death and differentiation, 16: 1030-1039, 2009
- Heim R and Tsien RY, Engineering green fluorescent protein for improved brightness, longer wavelengths and fluorescence resonance energy transfer. Current biology, 6: 178-182, 1996
- Miyawaki A, et al. Fluorescent indicators for Ca2+ based on green fluorescent proteins and calmodulin. Nature, 388: 882-887, 1997
- Piston DW and Kremers GJ, Fluorescent protein FRET: the good, the bad and the ugly. Trends in biochemical sciences, 32: 407-414, 2007
- Miyawaki A, Development of probes for cellular functions using fluorescent proteins and fluorescence resonance energy transfer. Annual review of biochemistry, 80: 357-373, 2011
- Hochreiter B, et al. Fluorescent proteins as genetically encoded FRET biosensors in life sciences. Sensors (Basel, Switzerland), 15: 26281-26314, 2015
- Hu CD, et al. Visualization of interactions among bZIP and Rel family proteins in living cells using bimolecular fluorescence complementation. Molecular cell, 9: 789-798, 2002
- Hu CD and Kerppola TK, Simultaneous visualization of multiple protein interactions in living cells using multicolor fluorescence complementation analysis. Nature biotechnology, 21: 539-545, 2003
- Alford SC, et al. Dimerization-dependent green and yellow fluorescent proteins. ACS Synth Biol, 1: 569-575, 2012
- Ding Y, et al. Ratiometric biosensors based on dimerization-dependent fluorescent protein exchange. Nature methods, 12: 195-198, 2015
- Lundholt BK, et al. A simple cell-based HTS assay system to screen for inhibitors of p53-Hdm2 protein-protein interactions. Assay and drug development technologies, 4: 679-688, 2006
- Shen K, et al. CaMKIIbeta functions as an F-actin targeting module that localizes CaMKIIalpha/beta heterooligomers to dendritic spines. Neuron, 21: 593-606, 1998
- Miller CL, et al. Virus-derived platforms for visualizing protein associations inside cells. Molecular & cellular proteomics, 6: 1027-1038, 2007
- Zolghadr K, et al. A fluorescent two-hybrid assay for direct visualization of protein interactions in living cells. Molecular & cellular proteomics, 7: 2279-2287, 2008
- Herce HD, et al. Visualization and targeted disruption of protein interactions in living cells. Nature communications, 4: 2660, 2013
- Lee S, et al. Small-molecule-based nanoassemblies as inducible nanoprobes for monitoring dynamic molecular interactions inside live cells. Angewandte Chemie (International ed. in English), 50: 8709-8713, 2011
- Hyman AA, et al. Liquid-liquid phase separation in biology. Annual review of cell and developmental biology, 30: 39-58, 2014
- Berry J, et al. RNA transcription modulates phase transition-driven nuclear body assembly. Proceedings of the National Academy of Sciences of the United States of America, 112: E5237-5245, 2015
- Li P, et al. Phase transitions in the assembly of multivalent signalling proteins. Nature, 483: 336-340, 2012
- Saio T, et al. PCS-based structure determination of protein-protein complexes. Journal of biomolecular NMR, 46: 271-280, 2010
- Ren J, et al. Structural and biochemical insights into the homotypic PB1-PB1 complex between PKCzeta and p62. Science China. Life sciences, 57: 69-80, 2014
- Karasawa S, et al. A green-emitting fluorescent protein from Galaxeidae coral and its monomeric version for use in fluorescent labeling. The Journal of biological chemistry, 278: 34167-34171, 2003
- Watanabe T, et al. Genetic visualization of protein interactions harnessing liquid phase transitions. Scientific Reports, 7: 46380, 2017
- Fluoppi (Fluorescent based technology detecting Protein-Protein Interactions)
[概要・緑色] [赤色] [製品リスト] - タンパク質間相互作用解析キット「CoralHue™ Fluo-chase Kit」[概要] [製品リスト]
- Fucci (Fluorescent Ubiquitination-based Cell Cycle Indicator) [概要] [製品リスト]
- 細胞小器官標識用発現プラスミド [概要] [製品リスト]
- 蛍光タンパク質抗体製品 [概要] [製品リスト]
- CoralHue™ Basic fluorescent proteins [概要]
- Keima-Red [概要] [Keima-Redによるマイトファジーの検出] [製品リスト]
- Kaede [概要] [製品リスト]
- Azami-Green [概要] [製品リスト]
- Kusabira-Orange [概要] [製品リスト]
- Midoriishi-Cyan [概要] [製品リスト]
- Dronpa-Green [概要] [製品リスト]
- Kikume Green-Red [概要] [製品リスト]
- Umikinoko-Green [概要] [製品リスト]
- Kusabira-Cyan [製品詳細]
- CoralHue™ ベクター全DNA配列