東京大学 深田 吉孝 先生より 『体内時計を制御するChronocodeという新概念』
体内時計を制御するChronocodeという新概念:
時計タンパク質の修飾リズムが織りなす時刻形成機構
 深田 吉孝 先生
深田 吉孝 先生
東京大学大学院理学系研究科生物科学専攻・教授
【略歴】
1978年 3月 京都大学理学部卒業
1983年 3月 同大学大学院理学研究科修了 博士(理学)
1983年 4月 札幌医科大学医学部第一生化学教室 助手
1986年 9月 京都大学理学部生物物理学教室 助手
1993年 5月 東京大学教養学部 基礎科学科第一 助教授
1995年 11月〜 東京大学大学院理学系研究科生物化学専攻 教授
1996年 4月〜1997年 3月 国立遺伝学研究所集団遺伝研究系客員研究部門 教授併任
1996年 4月〜2001年 3月 JST戦略的基礎研究CREST「脳を知る」 研究代表者
2007年 4月〜2010年 3月 日本学術振興会学術システム研究センター生物系科学専門調査班 専門研究員
1.体内時計と研究の歴史
『時間生物学』Chronobiologyとは、時間の関数として変化する生物現象の中でも、特に一定の周期性を示すリズミックな変化を扱う学問分野を指します。老化のように、時間と共に一方向に変化する現象は直接的な研究対象に含まれません。生物リズムの多くは細胞自律的な体内時計が支配すると考えられ、なかでも「概日時計」とは、地球の自転と公転で規定される1日(=24時間)周期に近い概日性の(概ね1日1サイクルの)リズムを支配する時計機構を指します。 動物においては、活動・睡眠・体温・血圧・内分泌・自律神経機能など、多くの生理機能が概日リズムを示し、概日時計の影響を受けない生理機能はないと言われるほど広範な影響を持っています。
1930年台に提唱された生物時計という概念に基づいて、1971年、ショウジョウバエの羽化リズムが異常を示すper変異体が単離され、その13年後のper遺伝子のクローニングによって時計遺伝子が初めて姿を現しました。その後、爆発的な時計遺伝子クローニングの時代に入りましたが、約半数の時計遺伝子を我が国の研究者が発見したという事実は特筆すべきでしょう。概日時計は、環境の明暗サイクルに同調するという重要な特性を持つため、概日リズムの研究は、時間生物学と光生物学という二つの学問分野の内包として発展しました(図1)。我が国では、この二つの分野に優れた研究者を数多く輩出し、両分野の融合した土壌において概日時計の研究が大きく開花したといえます。
図1.時間生物学と光生物学の内包として花開いた概日リズムの研究分野
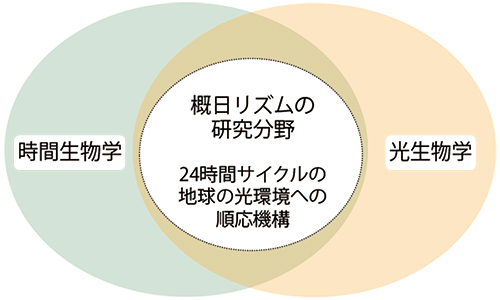
生物の概日時計において、24時間周期の明暗サイクルに同調できない時計はない。概日時計は次の一日のサイクルを予想できるという有利性を生物にもたらした。
2.時計遺伝子の転写フィードバック制御と3つの謎
時計遺伝子の実体が解き明かされる過程で、「時計遺伝子の翻訳産物が自らの転写を負に制御する」というフィードバック機構が時計発振の仕組みとして提唱されました。ここで常に注目されてきたのが、概日時計の基本的な性質にかかわる三つの謎です。まず第1に、24時間という極めて長いサイクルがどうして何十日も安定に振動し続けるのか、この頑強性はロバストネスという言葉で表現されます。これに対比して、時計は外部シグナルに対しては極めて柔軟に応答して位相がシフトするが、その分子メカニズムは何か、これが第2の謎です。第3の謎は、約24時間という時計周期が温度によって大きく変化しないように、周期の温度依存性がいかに抑えられているのか、という疑問です。この特性は、時計周期の「温度補償性」と呼ばれます。
3.末梢時計という新しい概念
これら三つの謎を解くための鍵を与える二つの新しい概念が2000年前後に現れました。一つは末梢時計の概念です。つまり、個体は無数の細胞時計から構成されており、それらの階層構造とカップリングから個体の時計システムが成り立っているという概念です。概日時計機構の中枢は視床下部の視交叉上核に局在する事から、従来、時計機能はニューロンの特異的な細胞機能の一つであると考えられてきましたが、培養細胞をはじめ体内の様々な器官や組織において時計遺伝子に発現リズムが見られ、その振動メカニズムは視交叉上核の中枢時計と基本的には同一であることが判明しました。その結果、時計機構はほぼ全ての細胞で作動している普遍的な細胞機能であることが分かり、視交叉上核の中枢時計に対比させて末梢時計という言葉が生まれました。生体は非常に多数の時計細胞から成り立ち、その「カップリング」が時計の安定性の謎を解く鍵であると考えられたのです。
4.リン酸化リズムが振動を生み出すという新しい概念
もう一つの新しい概念は、時計タンパク質のリン酸化リズムこそが時計発振に重要であるというシアノバクテリアでの発見に基づくものです。時計発振の中心メカニズムと考えられていた時計遺伝子の転写リズムに対して、これまでに提唱された多様な生物種の時計発振メカニズムは、全て図2に示したように、共役した二つのループで構成されます。従来、この左側の転写翻訳に基づくフィードバックループこそがリズム発振の中心的な機構と考えられ、これと並行して観察される時計タンパク質のリン酸化リズム(右側のループ)は、24時間という長い周期性を作りだすためのサブループあるいは安定化のための補助ル−プと位置づけられていました。
図2.時計タンパク質のリン酸化リズムの意義
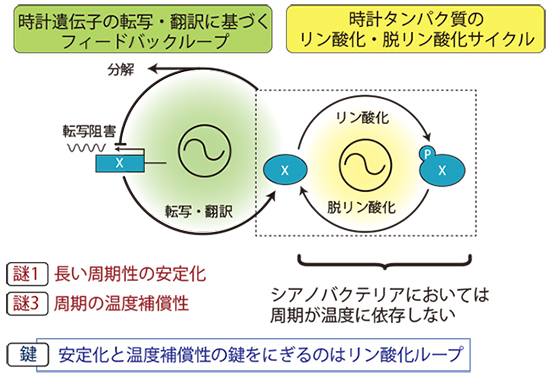
シアノバクテリアからヒトにいたる全ての時計振動機構は転写フィールドバックループとリン酸化サイクルで構成される。
ところが、シアノバクテリアのKaiCタンパク質の自己リン酸化と脱リン酸化のサイクルだけで概日時計が発振し得る事が示され、リン酸化リズムが時計発振の中心メカニズムになり得ることが分かりました。現在のところ、このようなメカニズムが動物の多くの時計機構にも当てはまるのか不明ですが、ここで強調すべきポイントは、KaiCの自己リン酸化リズムに基づく分子時計の周期が温度補償性を示す事です。概日時計の第3の謎である時計周期の温度補償性の仕組みは、動物細胞においてもリン酸化サイクルに重要な鍵が隠されている可能性があり、これまでに考えられているリズム安定化という意義に加えて、リン酸化サイクルの複雑な調節機構を解き明かす事が、今後の最重要課題の一つと言えます。
私共は最近、時計細胞の間の局所的カップリングにはTGF-/activin受容体キナーゼ(ALK)[文献1]やCa2+/カルモジュリン依存的なキナーゼ(CaMKII)[文献2]が重要な役割を果たすことを見出しました。時計タンパク質のリン酸化は、時計の階層構造の形成にも共通して登場するようです。
5.時計タンパク質の翻訳後修飾ネットワーク:Chronocodeとは?
図3に示したように、ほぼ全ての時計タンパク質がリン酸化され、その多くのリン酸化レベルが概日リズムを示します。多くの時計タンパク質には、リン酸化だけでなくユビキチン化、アセチル化、SUMO化、メチル化など、多彩な翻訳後修飾が見つかり、それぞれ概日時計の周期や位相の調節に重要であることが示されてきています。
図3.Chronocode: 時計タンパク質の翻訳後修飾の時空間制御
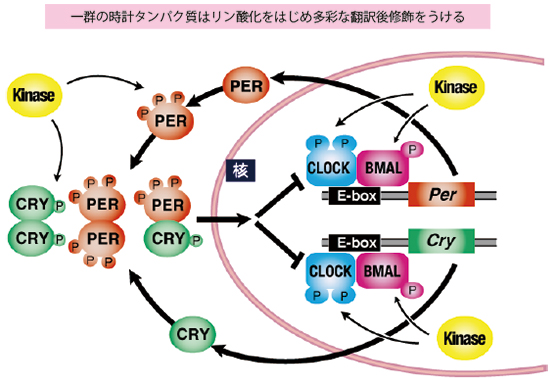
ほとんどすべての時計タンパク質は複数のキナーゼによってリズミックなリン酸化制御を受ける。
私共は最近、時計タンパク質CRYをユビキチン化する二種類のE3リガーゼを見つけ、これらがCRYの分解促進と安定化という拮抗する作用をもたらすことを発見しました。これらのE3リガーゼは、それぞれ、時計振動を早く進めようとする作用と遅くしようとする作用をもち、両者を欠失すると時計の振動が不安定化して概日リズムが崩壊します[文献3]。たった一つの時計タンパク質のユビキチン化だけをみても、かなり複雑な制御ネットワークが作動しているようです。
このように見てくると、リン酸化を代表とする翻訳後修飾の複雑な組み合わせによって一群の時計タンパク質の「存在量」、「細胞内分布」、そして「機能」が調節され、その結果として時計が安定に時を刻んでいると考えられます。つまり時計タンパク質の「各アミノ酸残基の修飾状態」によって一日の時刻は定義されているという考え方に基づき、時計タンパク質における翻訳後修飾の時空間制御に焦点を絞った『Chronocode』という概念をここで提唱したいと思います。私のこの造語は、ある時刻のスナップショットを見た場合に、修飾パターンがコードされているように見える、という受動的な言葉ではありません。むしろ、時(chrono)と共に推移する修飾状態の組み合わせ(code)が、正確な時を刻む能動的な駆動力であると考えます。複雑な修飾状態の組み合わせを正確に把握することにより、時計振動のダイナミズムを理解する事が可能になるという考え方です。例えばヒトPer2遺伝子の一塩基置換によるアミノ酸の点突然変異により、睡眠が大きく位相前進し、深夜に目覚めて夕方は眠くて起きていられない、という家族性の睡眠相異常が見つかっており、このアミノ酸はPER2タンパク質のリン酸化部位である事からも、リン酸化の時空間制御の重要性は明らかです。
6.時計タンパク質抗体を用いてChronocode解明へ
このようなChronocode研究において、一群の時計タンパク質を高感度に検出できるツールは何よりも心強い武器になります。通常のタンパク質抗体で翻訳後修飾を検出することは難しいことが多いですが、それでもリン酸化に伴うバンドシフトやユビキチン化タンパク質を高感度に検出できる抗体があれば、実験を次のステップに進める大きなきっかけを与えてくれます。私共は高感度の抗CLOCKモノクローン抗体やBMAL1モノクローン抗体を作製し[文献4]、これらの転写因子複合体がJNK[文献5]やCaMKII[文献2]によってリン酸化されて機能制御されることを示してきました。CLOCK抗体を用いたChIP解析では、CLOCK(とBMAL1との複合体)によりE-box依存的に転写活性化される約8,000箇所ものCLOCK結合部位をゲノムワイドに見つける事ができました[文献6]。また、CRY抗体やそのリン酸化抗体を作出する事によって、CRY2がDYRK1AとGSK3によって段階的にリン酸化され分解に導かれる事を見出しました[文献7,8]。このような定序リン酸化は他の研究分野において珍しい例ではありませんが、Chronocodeの概念を基に、抗体を一つのツールとして活用する事によって翻訳後修飾の時空間制御の全体像を解明できれば、そう遠くないうちに時計研究のルネッサンスを迎えることができると考えています。
文献
1. Kon et al. (2008) Nature Cell Biol. 10: 1463.
2. Kon et al. (2014) Genes Dev. 28: 1101.
3. Hirano et al. (2013) Cell 152: 1106
4. Yoshitane et al. (2009) Mol. Cell. Biol. 29: 3675.
5. Yoshitane, Honma et al. (2012) EMBO Rep. 13: 455.
6. Yoshitane et al. (2014) Mol. Cell. Biol. 34: 1776.
7. Kurabayashi et al. (2010) Mol. Cell. Biol. 30: 1757.
8. Hirano et al. (2014) Mol. Cell. Biol. 34: 4464.